多様化する次世代シークエンスの現状

※本記事は2012年12月号BioGARAGEに掲載したものです。次世代シークエンスの受託解析につきましては [email protected] までお問い合わせください。
2005年10月に454LifeSciences社から世界初の次世代シークエンサー(NGS)GenomeSequencerSystemGS20が発売されてから7年が経った。その間にNGSは世代交代を繰り返し、ハイスループット化を図る機種、またロースループットだが低コスト、短期間で解析可能な機種など多様な選択肢が生まれている。一方、受託解析サービスも展開され、ヘビーユーザーでなければ機器を買うよりもプロに委託した方が良いという状況にもなりつつある。
本特集では、NGSのヘビーユーザーではない研究者の方々を対象とし、現時点で市場に並ぶシステムの原理と特徴を紹介する。機種ごとに適したアプリケー ションなどを把握し、自らの研究にNGSを取り入れる際の参考にしていただければ幸いである。
各社の解析原理NGSの登場前は、シークエンスといえばサンガー法によるキャピラリーシークエンスが主流であった。一方、NGSでは各社がそれぞれ異なる原理での解析を行なっており、その情報はなかなかひとつにまとまっていない。そこで、簡易ながら、まずは各社がとる解析原理を眺めてみよう。なお、ここでは現在主流で使われているイルミナ、ロシュ、ライフテクノロジーズ(旧アプライドバイオシステムズ)が採用している技術に加え、1分子シークエンスと超ロングリードを売 りにして2011年に発売されたパシフィックバイオサイエンスの技術を取り上げる。
イルミナ
ブリッジPCR+ステップ・バイ・ステップの蛍光検出
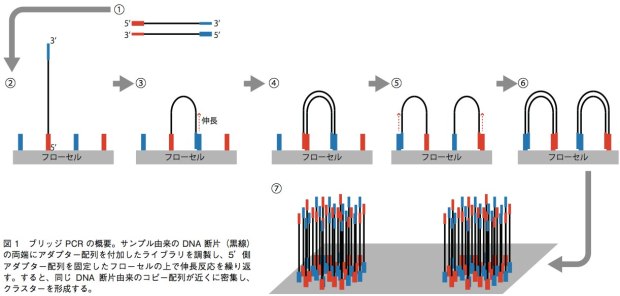
GenomeAnalyzer(GA)IIxやHiSeq、MiSeqでは、ブリッジPCRという方法を用いて、1本鎖DNA断片のクラスターをフ ローセル表面に作る。解析の際は、まずサンプルから得たDNA断片の両端に、2種類のアダプター配列(アダプター1、2)を連結させる。それを1本鎖にして、5’末端(アダプター1の側とする)をフローセル上に固定する。フローセル上には、あらかじめアダプター1、2と相補的に結合するプライマーが高密度に配置されており、サンプルの1本鎖DNAはアダプター2の側でこのプライマーと相補的に結合することになる(橋がかかったような構造になる)。この状態でDNAポリメラーゼによる伸長反応のあとで変性させると、フローセル上にはアダプター1側で結合した1本鎖と、アダプター2側で結合した1本鎖ができあがる。この反応を繰り返すことで、狭い面積の中で1本鎖DNAを固定しながら増幅することができる(図1)。これを鋳型として、配列解析を行うのだ。塩基配列の解析には、蛍光標識したdNTPの取り込みを蛍光顕微鏡によって解析する。このdNTPは3’末端がブロックされており、1回の伸長反応で1塩基しか伸ばせない。そのため、1塩基ごとにどのdNTPが取り込まれたかを観察し、蛍光物質とブロックを外して次の伸長反応を行うというステップで、解析を進めていく。シングルリード法ではアダプター1の側からのみ、ペアエンド法ではアダプター1の側と2の側からそれぞれ最大で150bpを解析可能となっている。
ロシュ
エマルジョンPCR+パイロシークエンス
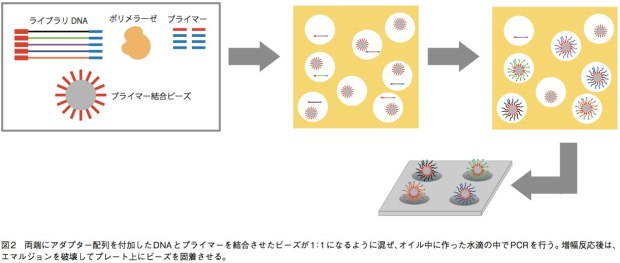
GS(Genome Sequencer) FLX+、GSjuniorでは、エマルジョンPCRによりDNA断片を増幅してから配列解析を行う。まず、サンプルから得て断片化したDNAの両端にアダプター(1、2とする)を結合させ、1本鎖にする。あるいは、リンカーDNAを挟んで断片の両端を連結して環状化し、その後サンプル配列の途中で切断してからアダプターを結合させる(mate-pair用ライブラリ)。そして、アダプターと相補的な短いDNAが結合したキャプチャービーズと、サンプルの1本鎖DNAとが1:1で結合するように混合し、増幅試薬とともに油中水滴エマルジョンに内包させる。これにより、オイル中にビーズひとつとDNA断片ひとつだけを持つマイクロリアクターを形成するのだ。こうして、各DNA断片は他の配列が混ざることなく、ビーズ上で数百万コピーにまで増幅される。そしてエマルジョンを破壊してビーズを濃縮し、ビーズひとつが収まる穴が無数に開いたピコタイタープレート上に載せて配列解析を行う(図2)。プレート上ではアダプターに相補的なプライマーから、DNAポリメラーゼによる伸長反応が行われる。この際、伸長のための材料としてdATPのみを添加した反応、dGTPのみを添加した反応……というようにdNTPをひとつずつ入れ替えていく。伸長反応が起こればピロリン酸が遊離するので、ルシフェラーゼによる発光反応で検出できる。この技術は、リード長がGS FLX+で最大1000塩基、juniorでも400塩基と他の機種と比べて長い。そのため、ロシュの装置はde novoシークエンスに強みを持っている。
ライフテクノロジーズ
エマルジョンPCR+2baseエンコーディング
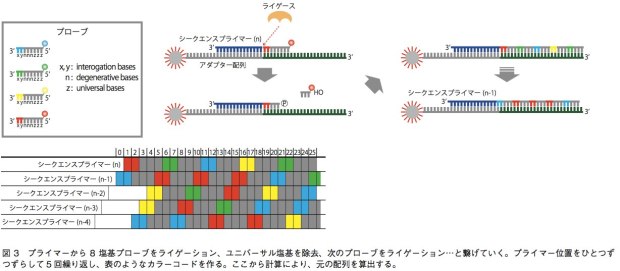
5500SOLiDシステムでは、配列解析のために用いられる増幅ステップは前項で書いたエマルジョンPCRと同様である。一方、塩基配列解析に関しては、DNAポリメラーゼを用いずにライゲーション反応を利用するという点で、他の技術と比べて大きく異なる。この技術では、A、T、G、Cいずれかが5つ並んだ後に、ユニバーサル塩基(どの塩基とも相補鎖を作れる)が3つ並んだ8塩基プローブを用いる。プローブにとって重要なのは、このうちの3’端の2塩基であり、あとの6塩基は鋳型DNA鎖と結合するためのみに使われている。プローブは、3’端の2塩基の配列に応じて、4色の蛍光色素で5’端が標識されている。TA、CG、GC、ATの場合は赤、AC、CA、GT、TGの場合は緑、AA、CC、GG、TTは青、GA、TC、AG、CTはオレンジといった具合だ。まず、エマルジョンPCR後にスライドガラス上に移されたビーズ結合DNA断片に対し、5’端がちょうどアダプター配列の末端にあたるようなシークエンスプライマーをあてる。そこにプローブを混ぜてライゲーションを行うと、サンプルDNAと相補的な配列を持つプローブがプライマーに結合されることになる。すると、蛍光の色によって、3’端の2塩基がどのパターンなのかが絞り込まれる(例えば赤だったら、2塩基はTA、CG、GC、ATのいずれかになる)。そしてユニバーサル塩基と蛍光色素を除き、次のライゲーション反応を起こす。これを繰り返すと、4パターンのうちどれか2塩基があり、次の3塩基は不明、次の2塩基も4パターンのいずれか、次の3塩基は不明……というような情報が得られる。これを、シークエンスプライマーの位置を1塩基ずつずらして5回繰り返し、得られた情報の全てに矛盾しない元の塩基配列を計算によって導くのである(図3)。この技術の強みは、シングルリードの中で同一塩基について2回情報を得ることだ。例えば1番目の塩基について、最初のプライマーによる反応系と、1塩基ずらしたプライマーによる反応系で情報が得られる。これにより、SNPや1、2塩基のin/delと、エラーとの区別を行いやすく、リシークエンスに向いている。
エマルジョンPCR+pH変化検出
IonTorrentテクノロジーは、SOLiDシステムと比べると新しい技術で、現在市販されている中で唯一、解析に光を用いず、半導体による電位検出によって塩基を判定するシステムだ。この技術の原理はロシュのエマルジョンPCR+パイロシークエンスとよく似ているが、配列解析の際に伸長した塩基を検出する方法が異なっている。塩基の伸長反応の際、ピロリン酸だけでなく水素イオン(H+)が遊離する。IonTorrentテクノロジーでは、このH+が半導体に与える電位変化を検出している。この技術のもうひとつの特徴が、シークエンス反応を行う半導体内で、データ処理も同時に行えることだ。これにより、生データ処理のためのコンピューターを別途用意する必要がなくなっている。また、検出に光学系が不要なことも合わせ、装置のコストが安く抑えられている。
パシフィックバイオサイエンス
1分子シークエンシング
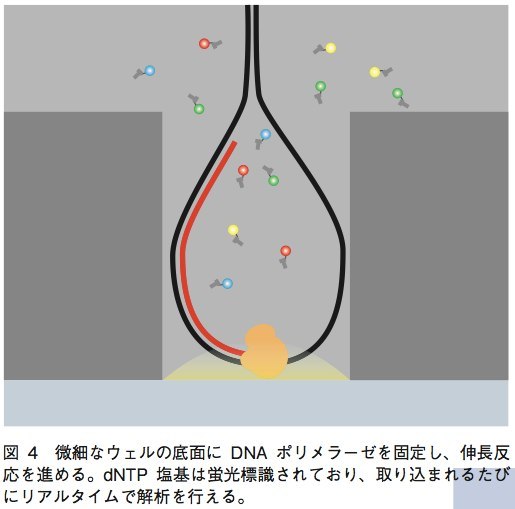
PacBioRSは現状で唯一、PCRによるサンプルDNA断片の増幅を行わない1分子シークエンサーである。シークエンス反応は、直径43.5μm、深さ32.8μmのウェル中で行われる。サンプルのDNA断片の両端にループ配列を結合させ、ダンベル状になったものを鋳型として用いる。この鋳型DNAを、ウェルの底面に固定されたDNAポリメラーゼに結合させ、伸長反応を行うのだ。この際、dNTPのリン酸基が蛍光色素で標識されており、伸長ごとに遊離する。この遊離蛍光を検出することにより、伸長反応を進めながらリアルタイムに塩基の解析ができるというのがPacBioの技術である(図4)。この技術の大きな特徴は、伸長される新規DNA鎖が通常のDNAと同じ構造を持つため安定性が高く、数kb〜数十kbものリード長を実現できることであ る。また、PCRを行わないために増幅時のバイアスやエラーを考慮する必要がなく、ライブラリ作製からDNA配列を得るまでの時間が短くて済むという利点もある。
NGSの性能を元に、適したアプリケーションを考える
NGSの用途は幅広い。ヒトなどゲノム決定済みの生物種の個体ゲノム全体を読むゲノムリシークエンス、エキソンなど一部領域に絞って読むターゲットリシークエンス、トランスクリプトーム、変異解析、smallRNAシークエンス、微生物の新規ゲノム解析、動植物の新規ゲノム解析、メタゲノム解析など、従来はマイクロアレイが担っていた役割も、NGSでの解析が可能になっている。これまで解説をしてきた各社の装置がどのようなアプリケーションに適しているか、次ページの表にまとめた。大まかに考えて重要になるのは、リード数とリード長、そして総解析塩基数だ。それぞれがどのようにアプリケーションの 適/不適に関わるのか、考えてみよう。
リード数
数百bしか読まないキャピラリーシークエンスと異なり、NGSは一気に数Mb〜数十Gbを解析する。そのため、ポリメラーゼによるエラーが0.001%でもあれば、読み間違いの塩基数は大きなものになる。そこで通常、同じ配列を数十回カバーするようにシークエンスを行う。もし装置のリード数が少なければ、仮にリード長が長かったとしても、限られた領域しか解析ができなくなる。例えば10万リードのシークエンサーで×20のカバレッジを作ろうとしたら、5000か所しか読めない計算になるのだ。 このように、リード数が少ないNGSは、限られた領域の解析に向いている。例えばエクソーム解析を含むターゲットリシークエンスやメタゲノム解析、ゲノムサイズの小さな生物のシークエンス等である。
リード長
リード長は、ひとつひとつのDNA断片に対するシークエンス反応で、どれだけの長さを解析するか、という値である。リード長が短いものは、得られた断片の配列同士を繋ぎ合わせるために、リファレンス配列が必要となる。そのため、denovoシークエンスにはリード長が長いシークエンサーの方が適している。また、繰り返し配列が多いゲノムの場合にも、リード長が長い方が良いだろう。パシフィックバイオサイエンスのPacBioRSは無類のリード長を誇る。今はまだエラー率が高いなどの難点もあるが、この点がクリアされれば他にない特徴を持ったシークエンサーとなるだろう。
総解析塩基数
ヒトなどサイズの大きなゲノムを解析する場合、当然シークエンサーにも大きな総解析塩基数を求めれる。イルミナのGAIIxやHiSeqが現在市場を最もリードしているのも、圧倒的な総解析塩基数と、ポリメラーゼによる伸長反応というスタンダードな解析原理を採用しているが故にヒトゲノムのリシークエンス等に強いためだろう。また、これらのNGSはフローセルを区切って異なるサンプルを同時解析できるため、総解析数が少なくて済むターゲットリシークエンス等についても多サンプルを処理できるという利点もある。ただし、1ランあたりのコストが高く、時間も長くかかるのがデメリットと言える。 最近では、MiSeqやライフテクノロジーズのIonシリーズ、ロシュのGSjuniorなど、総解析塩基数を抑え、アプリケーションを絞る代わりにコストが低く稼働時間が短いNGSが増えている。各種生物のゲノムデータベースが整備されてリファレンス配列が整い、エクソーム解析やトランスクリプトーム解析などを目的とする研究者が増えてきたからこその流れだろう。
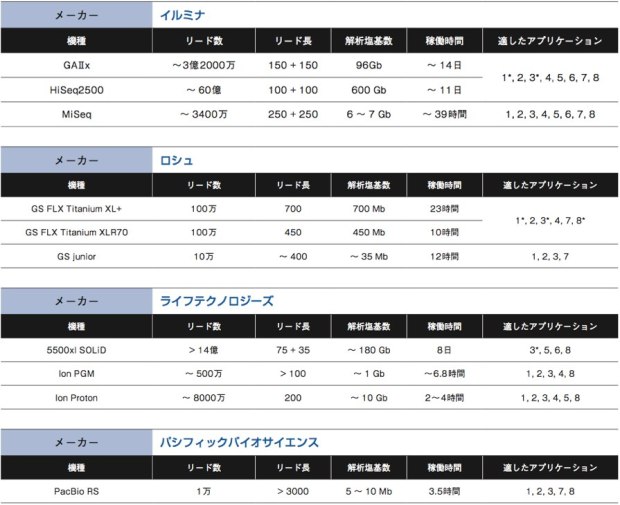
1 de novoバクテリア、プラスチド、微生物ゲノム
2 トランスクリプトーム解析
3 ターゲットリシークエンス
4 de novo 植物・動物ゲノム
5 ゲノムリシークエンス、転写量解析
6 変異解析
7 メタゲノム
8 その他(ChIP-Seq、small RNAシークエンス、メチル化解析など)
* 複数サンプルをまとめて解析しなければ、高コストとなる
解析前のサンプル・ライブラリ調製
次世代シークエンスを実施する際には、事前のライブラリ調製が必要となる。この調製ステップを細かく分けて見てみると、DNAの断片化、末端の平滑化、リン酸基付与、3’末端へのA付加、アダプター配列の付加、というようになっている。これらの反応を行うために必要な試薬がキットとして各社から発売されているので、基本的にはそれを利用するのが良いだろう。またこのステップの中で、断片化したDNAや調製したライブラリのクオリティチェックとして、純度や長さを確認することも重要だ。純度測定にはサーモフィッシャーサイエンティフィックのNanoDropなどの分光光度計を用いて280nm/260nmや260nm/230nmのOD比を確かめるのが良いだろう。長さの測定は電気泳動を行えばいいが、高い分解能がある方が良い。アジレント・テクノロジーのBioanalizerは、微量のサンプルから高精度に断片長を測定することができる。またパーキンエルマーのLabChip XTは断片長を精度高く測定した上で、目的サイズの断片だけを自動で精製することも可能だ。同シリーズのLabChip GXは96サンプルを同時に電気泳動にかけ、高いコントラストと分解能で泳動像を得られるので、アダプター配列の導入前後の分子量変化を見ることでクオリティチェックを行うこともできる。これらの工程をすべて自動化する装置もパーキンエルマー(NGS Expressなど)やベックマン・コールター (SPRIworks Fragment Library System)から発売されているので、大型プロジェクトなどで解析スケールが大規模になってきたら、導入を検討するのも手だろう。
加速し続けるNGS開発
ここまで、今すでに市場にあるNGSを俯瞰してきた。しかし、NGSはコンピューターの進歩を超える勢いで成長し、革新を起こし続けている。2012年10月に国内で発売されたライフテクノロジーズのIonProtonが発表されたのは同年1月。その直後の2月に、オックスフォード・ナノポア・テクノロジーズがPCのUSBポートに直接差し込む形のMiniONシークエンサーを発表し、世界に衝撃を与えた。なんとシークエンサーそのものが使い捨てで、900ドル程度で販売する予定だというのだ。今はまだ、いつ発売されるかは不明だが、近いうちに市場に現れると思われる。また本記事でも紹介しているパシフィックバイオサイエンスは、PC用半導体メーカーとして有名なインテルと共同で、より精度の高いシークエンサーを開発しているという。 これらの動き全てを把握し、自らの研究に最も適したシステムを選択し続けるのは、労力的にもコスト的にも難しいと感じる人も多いだろう。そのような場合、受託解析サービスの利用を考えるのもひとつの手だ。総解析塩基数の解説項目でも述べた通り、ハイスループットのNGSではフローセルを区切って使うことで、多サンプルをまとめて解析できる。解析対象がエクソームや微生物ゲノムなどの場合、受託企業では他のユーザーのサンプルとまとめて解析を行うことができるため、低コストで解析を委託することが可能だ。いずれにせよNGSは、初めて自らの研究に取り入れる研究者にとってはまだハードルの高い装置だろう。日本にはNGS現場の会というユーザーコミュニティもあるし、各種の受託解析企業もある。基礎的な知識を持った上で、そのようなプロの意見も聞きながら、新しい解析に踏み出していただきたい。本特集が、その一助になれば幸いである。
[参考文献]
Travis C. Glenn(2011)Field guide to next-generation DNA sequencers, Molecular Ecology Resources, 11, 7590769
Lin Liu et al.(2012)Comparison of Next-Generation Sequencing Systems, Journal of Biomedicine and Biotechnology, Article ID 251364
National Human Genome Reserch Institute, Advanced Sequencing Technology Award 2012, http://www.genome.gov/275550070